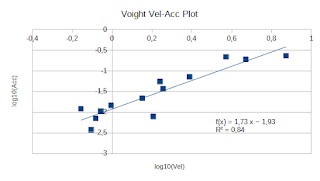
I dati sono estesi da ottobre 2023 a marzo 2024. La cosa interessante e' che il dataset di addestramento non comprende la accelerazione che e' iniziata il 3 di marzo e quindi la rete di fatto "non conosce" il trend di accelerazione
La differenza risulta nel fatto che LSTM si puo' usare per forecasting puro mentre per una post-analisi e' piu' conveniente BiLSTM
# Commented out IPython magic to ensure Python compatibility.
import numpy as np
import pandas as pd
from matplotlib import pyplot as plt
# %matplotlib inline
import seaborn as sns
import time
df = pd.read_csv('/content/ridotto.csv')
df['date'] = pd.to_datetime(df['Data'])
del df['Data']
columns = list(df.columns)
print(columns)
df_corr = df.corr()
df_corr
look_back = 20
sample_size = len(df) - look_back
past_size = int(sample_size*0.8)
future_size = sample_size - past_size +1
def make_dataset(raw_data, look_back=20):
_X = []
_y = []
for i in range(len(raw_data) - look_back):
_X.append(raw_data[i : i + look_back])
_y.append(raw_data[i + look_back])
_X = np.array(_X).reshape(len(_X), look_back, 1)
_y = np.array(_y).reshape(len(_y), 1)
return _X, _y
from sklearn import preprocessing
Xs = []
for i in range(len(columns)):
Xs.append(preprocessing.minmax_scale(df[columns[i]]))
Xs = np.array(Xs)
X_est, y_est = make_dataset(Xs[0], look_back=look_back)
X_temp, y_temp = make_dataset(Xs[1], look_back=look_back)
X_rain, y_rain = make_dataset(Xs[2], look_back=look_back)
X_con = np.concatenate([X_est, X_temp, X_rain], axis=2)
X = X_con
y = y_est
X.shape
y.shape
X_past = X[:past_size]
X_future = X[past_size-1:]
y_past = y[:past_size]
y_future = y[past_size-1:]
X_train = X_past
y_train = y_past
import tensorflow as tf
from tensorflow.keras.layers import Input, LSTM, Dense, BatchNormalization, Bidirectional
from tensorflow.keras.models import Model
from tensorflow.keras.optimizers import SGD, Adam
# LSTM
def create_LSTM_model():
input = Input(shape=(np.array(X_train).shape[1], np.array(X_train).shape[2]))
x = LSTM(64, return_sequences=True)(input)
x = BatchNormalization()(x)
x = LSTM(64)(x)
output = Dense(1, activation='relu')(x)
model = Model(input, output)
return model
# Bidirectional-LSTM
def create_BiLSTM_model():
input = Input(shape=(np.array(X_train).shape[1], np.array(X_train).shape[2]))
x = Bidirectional(LSTM(64, return_sequences=True))(input)
x = BatchNormalization()(x)
x = Bidirectional(LSTM(64))(x)
output = Dense(1, activation='relu')(x)
model = Model(input, output)
return model
model = create_BiLSTM_model()
model.summary()
model.compile(optimizer=Adam(learning_rate=0.0001), loss='mean_squared_error')
t1 = time.time()
history = model.fit(X_train, y_train, epochs = 350, batch_size = 64, verbose = 0)
t2 = time.time()
tt = t2 - t1
t_h = tt//3600
t_m = (tt - t_h*3600)//60
t_s = (tt - t_h*3600 - t_m*60)//1
print('Training Time : '+str(t_h)+' h, '+str(t_m)+' m, '+str(t_s)+' s')
predictions = model.predict(X_past)
future_predictions = model.predict(X_future)
plt.figure(figsize=(18, 9))
plt.plot(df['date'][look_back:], y, color="b", label="Real data")
plt.plot(df['date'][look_back:look_back + past_size], predictions, color="r", linestyle="dashed", label="prediction")
plt.plot(df['date'][-future_size:], future_predictions, color="g", linestyle="dashed", label="future_predisction")
plt.legend()
plt.show()